研究微生物多样性
保守亲缘关系,可变物种差异
原始数据,数据质控,高质量数据,聚类降噪,特征表和特征序列
二代分析可能只能进行种和科等级的鉴别,三代可以在属。
物种分类聚类热图
研究微生物多样性
保守亲缘关系,可变物种差异
原始数据,数据质控,高质量数据,聚类降噪,特征表和特征序列
二代分析可能只能进行种和科等级的鉴别,三代可以在属。
物种分类聚类热图
1,二代测序,三代测序的原理
2. 什么是CCS
chao 1和ACE是衡量物种丰富度的指标,和丰度、均匀度无关,对稀有的物种很敏感/
香农指数描述中的个体出现的紊乱和不确定性,不确定性越高,多样性就越高,香浓指数对群落的丰富度及稀有otu更敏感
聚类:DADA2
降噪:
ZOTUs 质心正确
ASV:质量值
分类:基于比对法、机器学习,两者结合
(条形图和热图)
多样性:a多样性和b多样性
a:Chao1(丰富度),Shannon指数(丰富度和稀有OTU),Simpson指数(对多样性即均匀度和丰富度,优势OTU更敏感)
b多样性:加权和非加权,基于独立OTU和基于系统发生树
相似性分析:分析组间差异是否大于组内差异,越接近1说明组间差异越大。
Adonis:置换多因素方差,分析不同分组因素对样品差异的解释度,置换检验来进行显著性分析,R2取值范围为【0,1】,越接近1说明分组因素对样品差异的解释度越高。
细菌微生物多样性和真菌微生物多样性
聚类:根据序列的相似性,聚类一些OTU
降噪:发现样品中真实的生物序列,模式不同
基于得到的特征表,进行各种分析
保守区-情缘关系
可变区-差异性
注意
一、研究对象和方法:
1. 细菌16S rDNA(1.5kb),保守区亲缘关系,可变区物种差异,
真菌ITS1,18S rDNA(1.8kb) 藻类、原生动物,
功能基因:固氮基因,硝化和反硝化。。。
2. 研究方法:
二代:短reads,短高变区V3+V4,V4等,只提供科或属级别分类;
三代:超长的测序读长,16S全长,在属和种水平上分类。
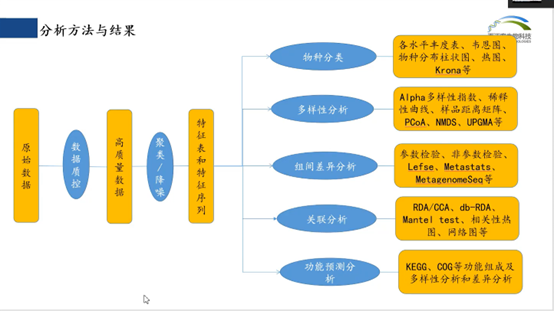
二、分析方法与结果:
多样性分析软件,物种分类,多样性分析,组间差异分析,关联分析,功能预测分析
1. 数据质控,聚类/降噪;
2. 物种分类:参考数据库,物种分布丰度,分布热图;
3. 多样性分析:
4. 组间差异分析
5. 相关性:RDA/CCA,相关性系数(Pearson,Spearman),网络图、表;
6. 功能预测分析:
微生物多样性
1.研究对象与方法:人粪便(疾病与微生物的关系)
2.研究方法:16s rDNA(细菌多样性)
3.分析结果:物种分析、多样性分析、组间差异分析、关联分析、功能预测分析。
4.数据质控:
4.1二代:PE reads拼接——引物序列识别——Tags过滤
4.2三代:CCS识别——引物序列识别——Tags过滤
5.聚类和降噪
聚类:OTU(序列相似性)
降噪:
6.物种分布:(歌物种组成)
7.多样性(A:内部 :辛普森多样性指数)——箱线图
B:样品距离(加权与非加权)
相似性差异:(ANOSIM):箱线图—PCA
8.组间差异:(相对丰度)
9.相关性与关联分析:
9.1排序分析:RDA/CCA
9.2相关系数分数
9.2网络分析()
10.功能预测分析
10.1
0716 学习
1.微生物研究对象:
2.研究方法(功能):细菌、真菌、功能基因
3.测序方法:二代测序--科属水平分类,短reads
三代测序--超长的,属/种水平鉴定
pacbio全长测序:序列扩增-加接头-识别ccs序列
4.分析方法和结果:
原始数据-聚类-特征表/序列
--》物种分类图
--》多样性分析
--》组间差异性分析
--》关联分析(物种与物种之间,物种与环境之间的关系)
--》功能预测分析(推测样品功能--重复以上4个分析作图)
数据质控(剔除低质量数据)-方法
聚类/降噪
聚类--基于序列相似性得到OTU,一个OTU中可能包含一些具有不同表型的菌株或物种的生物序列
降噪--ZOTUS 和 ASVs两种方法
实例解释:
物种分类:鉴定物种所在的等级
1.基于比对:blast/vsearch: 特征序列与参考数据库对比,找出一致性最高的
2.基于机器学习(贝叶斯、决策树)
3.比对结合机器:
物种分类分析:样品组成情况、各物种在不同样品中的丰度趋势变化--》热图的颜色变化
多样性分析:α多样性和β多样性
α多样性:单个样品物种丰富度以及物种多样性
衡量指标有:Chao 1和ACE,香浓、辛普森
β多样性分析:描述样品间的差异性
组间差异分析(比较组间和组内差异的大小):箱线图和PCoA图
相似性分析(ANOSIM):R值接近1,表示组间差异>组内差异,是比较好的。
Adonis置换多因素方差分析:R²越接近1说明分组因素对样品差异的解释度就越高。
组间差异分析(比较物种组与组之间的差异):常见的方法--T检验;方差分析;秩和
常见的问题:1.哪一种分析方法都有应用,多多尝试不同的方法;2.目前多样性分析的差异结果,一般还需要更多的重复和验证,才能最终加以说明。
相关性和关联分析: 主要研究的是 微生物与微生物、微生物与环境因子之间的相互作用,
有两类方法:
功能预测分析:PICRUSt2计算方法--利用标记基因数据和一个参考基因组数据库来预测微生物功能组成。