lefse找不同组间差异的方法
通常l选4
lefse找不同组间差异的方法
通常l选4
r越大显著性差异越大
丰度-差异
nmds,多维——一维
置信椭圆>5
等级丰度曲线,丰富度,均匀度
曲线平缓分布均匀,横坐标是丰富程度
otu聚类分析分析单元
组间差异分析,测序lefse方法。用已知预测未知。例如健康人与病人的差异,预测测样是否正常。
PC0A分析主成分分析降维,降噪差异不大去掉,去冗余
贡献率高,主成分可以反映原始信息
距离算法 独立otu,系统发生树
加权和丰度,非加权只看物种。
CAZy碳水化合物活性酶
TCDB膜转运蛋白数据库
PHI病原宿主互作数据库
P450单氧化酶CYPS
基因家族分析:聚类分析得到物种共有家族和特有家族,以及单拷贝直系同源基因
进化树:推断物种形成时间以及物种与其他物种之间进化关系。
基因家族扩张收缩,因为某类基因扩张导致相关功能强化或对特定生存环境适应性加强。cafe方法。
选择压力分析 ks同义突变率,氨基酸不变
ks非同义突变率,密码子改变氨基酸改变
研究微生物多样性
保守亲缘关系,可变物种差异
原始数据,数据质控,高质量数据,聚类降噪,特征表和特征序列
二代分析可能只能进行种和科等级的鉴别,三代可以在属。
物种分类聚类热图
1,二代测序,三代测序的原理
2. 什么是CCS
Ven n 图
展示的多个样品或多个组间共有和特有的物种或OTU 当out在某个样品序列条数为零则该OTU不存在于该样品
OTU个数分布_用于统计的不同样品 或不同组间的OTU数目
Seas-num _序列条数
chao 1和ACE是衡量物种丰富度的指标,和丰度、均匀度无关,对稀有的物种很敏感/
香农指数描述中的个体出现的紊乱和不确定性,不确定性越高,多样性就越高,香浓指数对群落的丰富度及稀有otu更敏感
聚类:DADA2
降噪:
ZOTUs 质心正确
ASV:质量值
分类:基于比对法、机器学习,两者结合
(条形图和热图)
多样性:a多样性和b多样性
a:Chao1(丰富度),Shannon指数(丰富度和稀有OTU),Simpson指数(对多样性即均匀度和丰富度,优势OTU更敏感)
b多样性:加权和非加权,基于独立OTU和基于系统发生树
相似性分析:分析组间差异是否大于组内差异,越接近1说明组间差异越大。
Adonis:置换多因素方差,分析不同分组因素对样品差异的解释度,置换检验来进行显著性分析,R2取值范围为【0,1】,越接近1说明分组因素对样品差异的解释度越高。
chao1 and Ace指数简单地反映出群落中物种的数量,而不表示群落中每个物种的丰度信息。Shannon and Simpon指数用于衡量群落多样性,受样品群落中物种丰度和物种均匀度(community evenness)的影响,chao1 and Ace指数用于衡量群落丰度。相同物种丰度的情况下,群落中各物种具有越大的均匀度,则认为群落具有越大的多样习性;chao1、 Ace、Shannon指数值越大,Simpon指数值越小,说明样品的物种多样性越高。
RDA/CCA分析,夹角角度“锐角正相关,钝角负相关;点点距离:距离越近,相似性越大
细菌微生物多样性和真菌微生物多样性
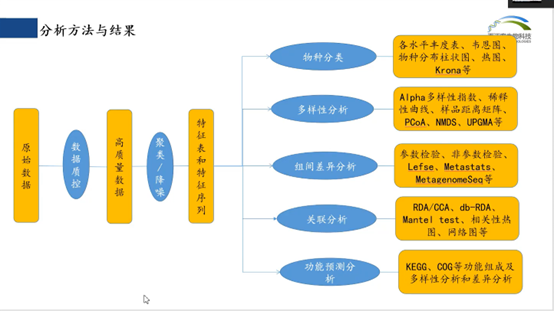
聚类:根据序列的相似性,聚类一些OTU
降噪:发现样品中真实的生物序列,模式不同
基于得到的特征表,进行各种分析
物种丰度聚类热图
颜色-丰度高低
保守区-情缘关系
可变区-差异性