九、微生物多样性数据上传:
1. 注册NCBI账号;
2. submit:原始测序数据,SRA(Sequence read archive)数据库,数据释放时间,项目名称和描述,*必填
3. 云平台上传,我的数据,fq文件。
九、微生物多样性数据上传:
1. 注册NCBI账号;
2. submit:原始测序数据,SRA(Sequence read archive)数据库,数据释放时间,项目名称和描述,*必填
3. 云平台上传,我的数据,fq文件。
八、微生物多样性结题报告:
1. cutomer_backup包括全部的分析结果,src和index.html是网页版结题报告,index.html打开;
2. 内容:概括,背景-细菌16s和真菌ITS
3. 每个OTU代表一个物种,
物种聚类热图-物种和样品聚类,
物种系统进化树-环形图是物种进化树,相同颜色代表相同门
beta多样性:在微生物多样性分析中,由于环境中的微生物复杂多样,各环境间物种的组成差异更为
剧烈,所以通常采用非加权方法进行分析。但如果要研究对照与实验处理组之间的关系采用非加权
分析比较不出明显差异,则更推荐加权分析方式。
NMDS:建议不分组时,样本数量不少于10个;多组样本时,每组样本数量不少于5个。
相关性:物种之间,点大小表示丰度,线粗细表示相关性,关键物种,物种稳定性。
七、物种聚类热图:
1. 结题报告-物种分类学-物种丰度热图,分析类型,分类水平,聚类分析选择-物种或样品,计算算法选择-距离算法(比较样地和样品的距离)和聚类算法(根据样地的距离的相似性进行聚类);
2. 物种丰度相对比例表,热图修改,配色方案和图标题。
六、PCA、PCoA、NMDS分析:
1. 结题报告-Beta多样性分析-PCA或PCoA,分析类型-样品间和组间,分类水平,提交;
2. 物种丰度相对比例表,PCA图,点之间距离反映差异大小,主标题-标号?
2. PCoA和NMDS:参数设置与PCA相似,只是多了一个距离算法选择,
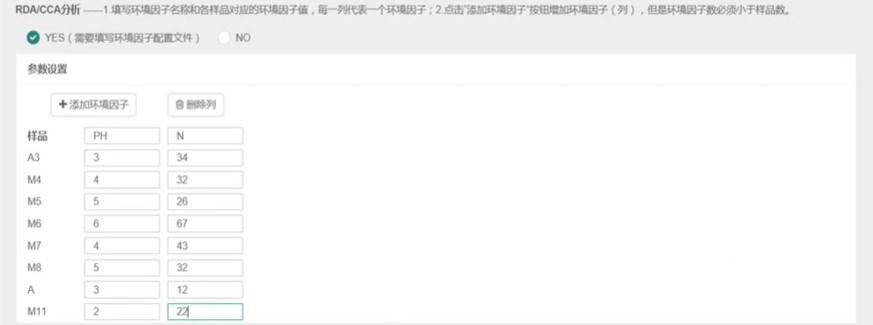
五 RDA/CCA:
1. 结题报告-相关性和关联分析,分析类型-样品间、组间,环境因子,环境因子-手动或上传,分类水平;
2. 物种丰度相对比例表,RDA图-蓝色环境因子,红色物种,点样品,修改图片,是否显示物种名称。
四. 物种分布柱状图:
1. 项目-报告-物种分类学,分析类型-样品间和组间,分类水平,丰度最高或比例;
2. 丰度相对比例表,柱状图;
3. 分析结果保存后,出现在分析记录里面
三. OTU的Venn图:
物种分类学分析,<5样品是venn图,>=5样品是花瓣图,图片下载、列表下载或一键下载,比如共有OTU情况,分析结果保存。
二. 微云数据上传:2种方法
1. 我的数据-个人数据,创建目录,上传文件小于1000M,原始数据,不是压缩数据;
2. FTP:大于1000M,邮件,FTP账号、密码,FTP客户端下载,ftp_upload文件夹.
一.. 微生物多样性分析平台微云操作步骤:
1. 综合选项:微生物多样性分析平台,项目名称、描述,未拼接双端数据(文件格式为fastq格式,双端测序数据文件名通常为XXX_1.fq、XXX_2.fq,文件两两配对缺一不可),已拼接单端数据(单端测序数据通常是以.fq 或 .fastq结尾)
2. 原始数据导入:文件夹(micr-diversity),文件. 填写引物数据,默认百迈克引物(normal),样品命名;
3. 样品数据合并:
4. 扩增子类型:分子标记类型-细菌16s, 真菌ITS;可变区,RDA/CCA分析,环境因子
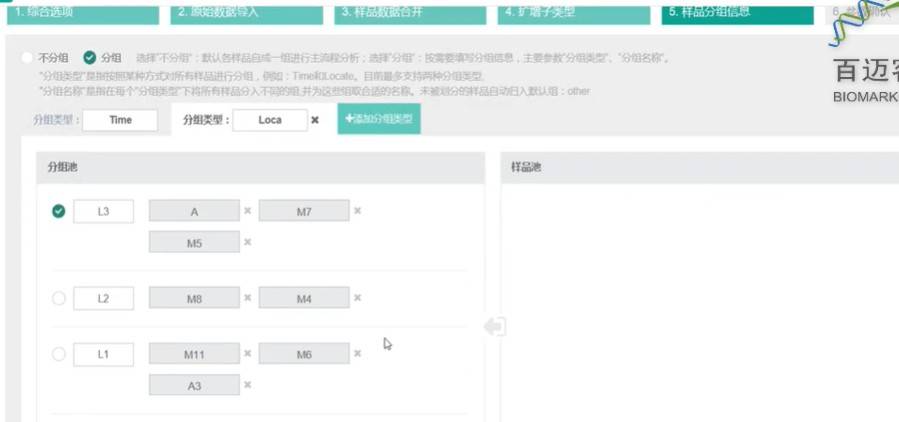
5. 样品分组信息:分组类型-时间,地点,具体分组,最多只有2种分组类型。
6. 参数确认:确认提交。
返回主界面,查看我的项目,模拟项目-极速体验项目。
4.扩增子类型
分子标记类型
细菌--16s
真菌--ITS
完整的分析过程,结题报告下载就行
绘制VENTU
个性化分析,绘制VENN图
样品间分析,样品选择(5个以及5个以内),5个以上是花瓣图
分析,
微生物多样性分析平台,
项目命名
项目描述()
导入数据(文件夹,或者单个样品)
引物填写,(逗号分隔)
样品命名
数据合并的选择
添加合并组
默认不原则参与合并样品
进行数据类型的选择(环境因子)
分组(依据分组,只能有两种分组方式)
确认参数,提交
总览,我的项目,进行标准分析
4.3.3物种丰度聚类热图
4.4 Alpha多样性分析
物种累积曲线:测序样本量与检测到的物种的数量的关系
4.5 Beta多样性分析:比较不同样品在物种多样性方面存在的相似程度
4.5.1 PCA分析
4.5.4UPGMA
Beta多样性箱型图
OTU代表每一个物种
各等级OTU,
7:52
丰富度:群落中物种组成的种类
曲线越平坦物种组成的均匀度越高:群落中各物种丰度的差别(eveness平摊均匀)
物种累积曲线:测序样本量与检测到物种数量之间的关系
横坐标:抽取样本量的增加
红色:总共物种的数量:上升代表群落中不断有新物种被发现
趋于平缓:物种不会随着样本量的增加而增加
绿色:抽取样本中共有的物种的数量,在一定范围内若加大样本量,若曲线下降反应样本中新发现的物种逐渐减少,利用物种累积曲线可判断样本量是否充足,若曲线急剧上升则表示样本量不足,需要增加样本量,若样本量足够曲线
绿色:共有物种
Beta多样性分析,不同样本在物种多样性方面存在的相似程度,利用四种算法计算样本间的距离,而获得样本间的一个值
不同OTU之间的距离具有远近之分的。
数据处理交叠代表共有
未经过拼接得数据类型选择双端,已拼接得单端。
结题报告:
index.html网页版结题报告
下机数据:双端测序的reads,拼接完成为tags
数据处理:PEreads拼接→Tags过滤→去除嵌合体→有效数据(有效tag/原始)
Tags→OTU聚类
物种注释与分类学分析
qiime软件系统进化树?
面积代表在该分支处样品的序列多少
等级丰度曲线
β多样性通常用非加权
pcoa环境因子之间的夹角为锐角说明两因子之间为正相关,反之
β多样性箱线图,纵坐标为组内样品的差异性,箱越扁,说明组内差异越小
组间差异显著性分析:
lefes
metastates分析:一般认为p值小于0.05说明有显著性差异
kegg代谢途径
cog功能预测
相关性分析:物种之间的相关性,可以找到环境中的关键物种,对我们研究环境中物种的稳定性有
物种丰度热图:
距离算法:比较样品间的距离
聚类算法:根据样品间的距离,对他们间的相似性进行聚类。
PCA/PCOA/NMDS分析: